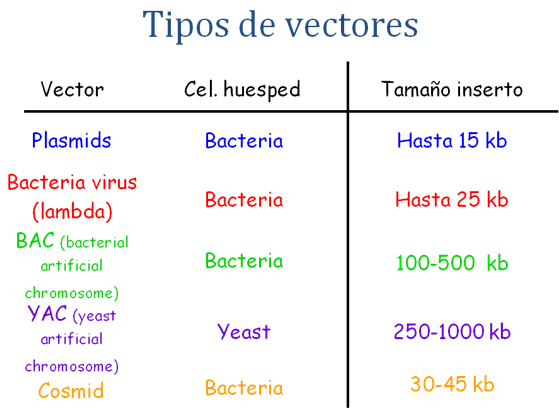
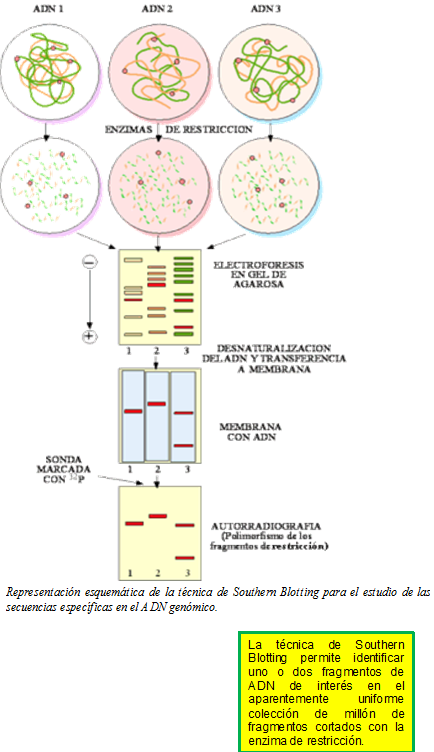
COMO SE REALIZA EL CONTROL GENETICO DE UN GEN BRCA
El BRCA1 es un gen localizado en el cromosoma 17q21, con herencia autosómica dominante y de alta penetrancia. Está conformado por 5,592 bases, un peso de 207 KDa y estructuralmente formado por 22 exones y 1,863 aminoácidos. Es un gen supresor de tumor, involucrado en la regulación del ciclo celular, reparación del ADN dañado, mantenimiento de la estabilidad genómica y regulación de la transcripción.2 Las mutaciones detectadas en este gen son: delaciones, inserciones, mutaciones puntuales, mutaciones sin sentido (codones de detención prematura), hipermetilación de la región promotora.
La mutación del BRCA 1 se da principalmente en grupos étnicos como los judíos Ashkenazi, grupos familiares de Noruega, Islandia, Suecia y Polonia. Dentro de las características sobresalientes en el cáncer de mama asociado a BRCA1 son: edad más temprana, mayor propensión a la multifocalidad y bilateralidad, alto índice mitótico y receptores estrógenos y progestágenos negativos.
Las mutaciones en los genes BRCA-1 y BRCA-2 son asociadas con un subgrupo de los cánceres del seno y de los ovarios. Estos genes tienen funciones diferentes dentro de las células. Así como los otros supresores de tumor, las mutaciones pueden ser heredadas o espontáneas.
Las personas que heredan mutaciones de BRCA-1 o BRCA-2 son más susceptibles al desarrollo del cáncer de los senos.
Los individuos que tienen una mutación BRCA corren un riesgo de desarrollar cáncer de los senos a un 80% (si viven a la edad de 85). El riesgo de desarrollar cáncer de los ovarios es 10-20% para las mutaciones BRCA-2 y 40-60% para las mutaciones BRCA-1. La presencia de estas mutaciones también puede aumentar el riesgo de cáncer de la próstata, del páncreas, del colon, y otros cánceres. El riesgo total de cualquier persona depende de los factores de riesgo genético y ambiental a los cuales se encuentran expuestos. Se estima que las mutaciones en BRCA-1 y BRCA-2 son asociadas con el 5-10% de todos los cánceres de los senos.
Estas mutaciones diferentes se pueden localizar dentro del mismo gen pero en diferentes localizaciones (heterogeneidad de locus) o en diferentes genes por completo (heterogeneidad alélica). Por ejemplo, la susceptibilidad al cáncer heredado de seno y de ovario tiene heterogeneidad tanto de locus como alélica. Más de 500 mutaciones diferentes han sido identificadas que pueden ocurrir en el gen BRCA1 en el cromosoma 17 y aumentar el riesgo de una mujer de desarrollar cáncer del seno. Y más de 300 mutaciones dispersas por todo el gen BRCA2 en el cromosoma 13 están asociadas con la susceptibilidad al cáncer heredado de seno y de ovario.
El Efecto de BRCA en la Sobrevivencia
Ya han existido varios estudios diseñados para determinar las diferencias en la sobrevivencia entre los individuos con cáncer que cargan la mutación BRCA y aquellos quienes desarrollan cáncer espontáneamente. Los resultados son algo contradictorios, posiblemente debido a las diferencias en el diseño del estudio y los factores tales como la definición de los controles (casos espontáneos) y los cargadores. Sin embargo, aunque los individuos que cargan la mutación BRCA tienen un peor pronóstico basado en las características de su cáncer, ellos parecen tener una tasa de sobrevivencia igual o mayor comparada con los pacientes de casos espontáneos. Se piensa que esto se debe a la respuesta de los tumores a la quimioterapia. La mayor susceptibilidad puede ser, en parte, por su mayor ritmo de proliferación. Los tumores también son más susceptibles a los tratamientos contra el cáncer tales como la radiación gamma, cisplatino, y mitomicina C porque estos tratamientos causan daños al ADN que normalmente serían reparadas por los productos BRCA funcionales. Si los genes BRCA están desactivados, la célula no puede reparar el ADN eficientemente y la célula muere. Las células no cancerosas en un individuo con mutación BRCA retienen una copia funcional del gen y pueden entonces reparar su ADN.
Sin embargo, un nuevo estudio indica que el gen está implicado asimismo en un proceso que no está relacionado con el cáncer. En "Cell" científicos del Dana-Farber Cancer Institute de Boston (Estados Unidos) explican que el BRCA1 desempeña un papel en cómo se expresa la información genética de la mujer.
Dado que las mujeres tienen dos cromosomas X, pero sólo uno de ellos está activado. En el nuevo estudio, los autores observaron que un defecto en el BRCA1 puede conducir a una alteración en la inactivación de uno de los cromosomas X en la mujer. Además, comprobaron que si se inhibe la función del BRCA1 normal, aumentan las probabilidades de que un gen localizado en el cromosoma X inactivado se active.
En opinión de los investigadores, el efecto de tener uno o dos cromosomas X funcionales se desconoce. Es algo que califican de "situación biológica inusual", aunque sugieren que las células que contienen dos cromosomas X activados tendrán más probabilidades de contener mayor número de genes expresados, escenario que tal vez estimule el crecimiento de células mamarias tumorales.
BIBLIOGRAFIA
ü http://www.cancerquest.org/index.cfm?page=341&lang=spanish